Natalia von Ranke, Wei Zhang, Philipp Anokin, Nicolas Hulscher, Kevin J. McKernan, Peter A. McCullough, John A. Catanzaro
Resumen
Antecedentes/Objetivos: Las vacunas sintéticas de ARNm han suscitado preocupación por la expresión prolongada de las proteínas espiga, la activación inmunitaria y los posibles efectos fuera de objetivo. Este estudio investiga las alteraciones transcriptómicas en individuos con efectos adversos de nueva aparición o cáncer tras la vacunación con ARNm COVID-19. Métodos: Se realizó la secuenciación de ARN masivo en sangre periférica de dos grupos de pacientes: individuos con efectos adversos no malignos de nueva aparición e individuos con diagnóstico reciente de cáncer tras la vacunación. Se utilizó un grupo de control de individuos sanos para la comparación. Se analizó la expresión génica diferencial mediante DESeq2 y se realizó un análisis de enriquecimiento de conjuntos de genes (GSEA) utilizando la base de datos MSigDB y conjuntos de genes personalizados. Resultados: Ambos grupos de pacientes vacunados mostraron una desregulación transcripcional generalizada. En el grupo de efectos adversos no malignos, el aumento de marcadores incluyó la disfunción mitocondrial, el estrés mediado por proteasomas, la inestabilidad transcriptómica y la inflamación sistémica. El grupo de cáncer exhibió distintivos adicionales de inestabilidad genómica y reprogramación epigenética. La desintegración mediada por sin sentido (NMD), el estrés ribosómico y la activación de MYC fueron prominentes en ambos grupos, mientras que la señalización inmunológica a través de TLR e interferones de tipo I fue particularmente elevada en los pacientes con cáncer. Conclusiones: Los perfiles transcriptómicos observados indican respuestas persistentes de estrés celular, disfunción mitocondrial y desregulación inmunitaria tras la exposición a vacunas de ARNm, potencialmente en individuos susceptibles. Las firmas moleculares compartidas y distintas en ambas cohortes demuestran mecanismos subyacentes que contribuyen a la sintomatología y las complicaciones postvacunales, incluida la oncogénesis y/o la progresión de la enfermedad maligna. Estos hallazgos subrayan la necesidad de una investigación más profunda sobre la seguridad a largo plazo de las vacunas de ARNm y la variabilidad de la respuesta del huésped.
2. Materiales y métodos […]
3. Resultados
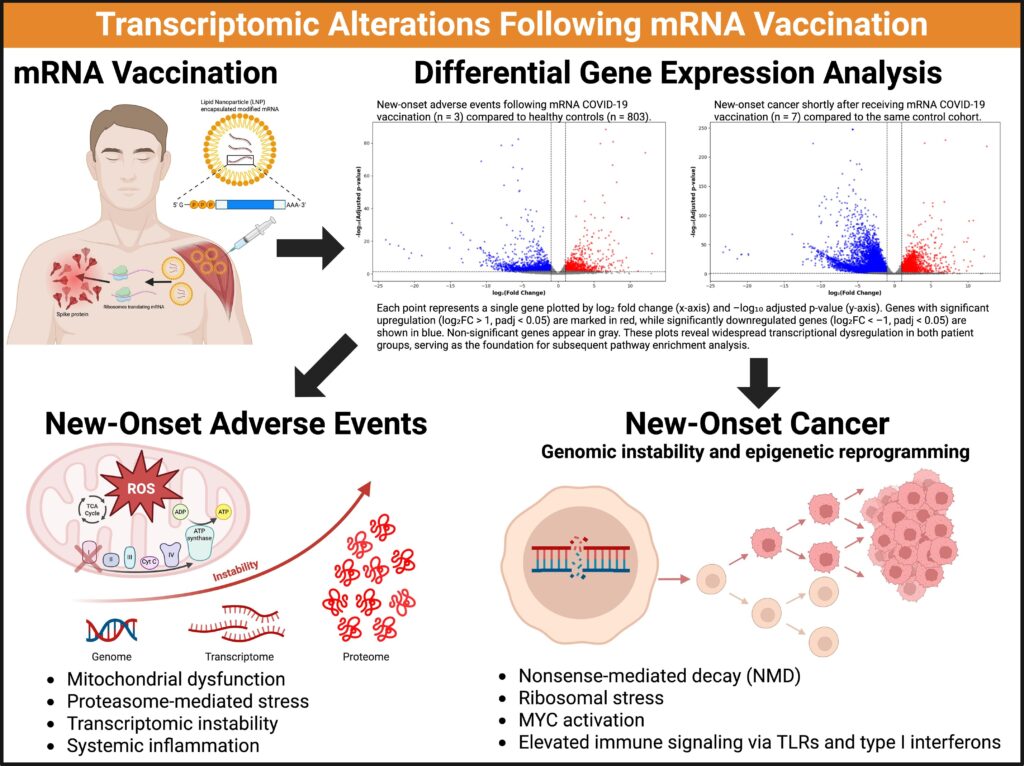
Para caracterizar el impacto transcripcional global de la vacunación ARNm en individuos con resultados adversos postvacunación, realizamos análisis diferenciales de expresión génica comparando cada grupo de pacientes con la cohorte de control sana. Como se muestra en la figura 1, los diagramas de volcán visualizan la distribución de los cambios en la expresión génica tanto para el grupo 1, síntomas postvacunación no malignos de nueva aparición (A), como para el grupo 2, diagnósticos de cáncer de nueva aparición (B). En ambos grupos, se observa un claro cambio transcriptómico, con cientos de genes que muestran una desregulación significativa. Notablemente, el grupo de cáncer exhibe una distribución más amplia de genes desregulados, incluyendo varios con magnitudes extremas de cambio de pliegues, lo que sugiere una supresión profunda de vías reguladoras clave.
El análisis de enriquecimiento de conjuntos de genes (GSEA) se realizó por separado para ambos grupos de estudio: Grupo 1, compuesto por individuos con efectos adversos de nueva aparición tras la vacunación con ARNm COVID-19, y Grupo 2, compuesto por individuos diagnosticados de cáncer de nueva aparición tras la vacunación, para caracterizar las alteraciones transcriptómicas asociadas a la exposición a la vacuna. El GSEA se llevó a cabo utilizando las colecciones predefinidas de conjuntos de genes MSigDB, y los conjuntos de genes enriquecidos se agruparon sistemáticamente en función de su relevancia para las vías moleculares distintivas de orden superior. Esta agrupación basada en vías permitió una interpretación integradora de las alteraciones transcriptómicas en todos los dominios de la biología de sistemas.
En el Grupo 1, los conjuntos de genes más enriquecidos se agruparon en seis categorías de marcadores clave: (1) Disfunción del transporte mitocondrial de electrones y especies reactivas del oxígeno (ROS), (2) Estrés por degradación de proteínas mediada por el proteasoma, (3) Inestabilidad transcriptómica y estrés traslacional, (4) Respuesta inflamatoria e inmunitaria sistémica, (5) Disfunción del endotelio y (6) Señalización proliferativa y control tumoral suprimido. Por el contrario, el Grupo 2 mostró un patrón de enriquecimiento distinto pero superpuesto. Los rasgos distintivos identificados en este grupo incluyen: (1) Inestabilidad transcriptómica y estrés traslacional, (2) Respuesta inflamatoria e inmunitaria sistémica, (3) Disfunción del endotelio, (4) Señalización proliferativa y control tumoral suprimido, y (5) Inestabilidad genómica y cambio epigenético. Como se muestra en la Tabla 1, múltiples conjuntos de genes dentro de cada categoría de sello distintivo demostraron fuertes puntuaciones de enriquecimiento (NES > 1,5 o < -1,5) y valores q de FDR estadísticamente significativos (FDR < 0,05), apoyando la presencia de una desregulación molecular coordinada. En la figura suplementaria S1 se proporcionan curvas de puntuación de enriquecimiento (ES) representativas para ilustrar la distribución de los rangos de genes que contribuyen a cada sello distintivo.
Para explorar más a fondo el panorama molecular asociado a las alteraciones transcriptómicas inducidas por la vacuna, se realizó un análisis de redes de interacción proteína-proteína (IPP) para los genes más significativamente desregulados en cada grupo de estudio. Como se muestra en la figura 2, el mapa PPI para el grupo 1 (individuos con efectos adversos de nueva aparición tras la vacunación) y la figura 3 muestra el mapa PPI para el grupo 2 (individuos con cáncer de nueva aparición tras la vacunación).
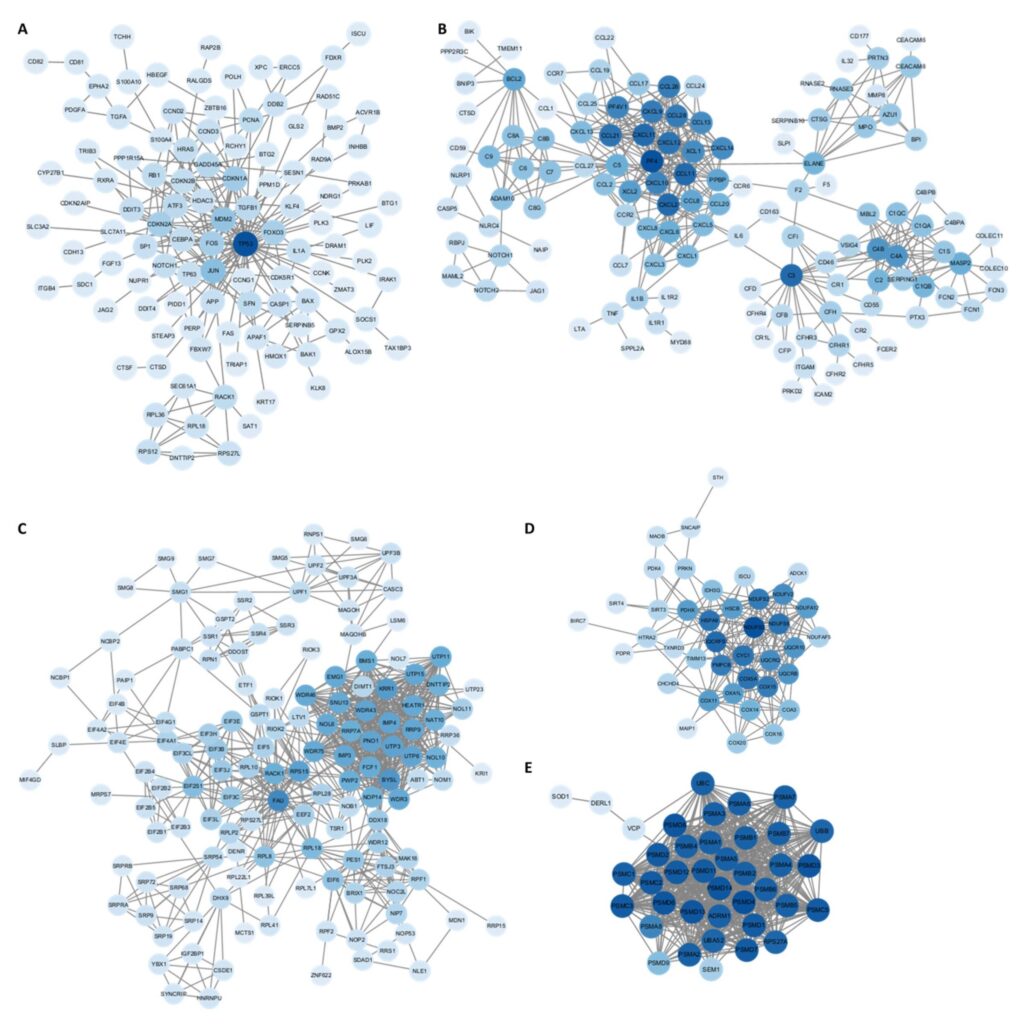
Figura 2. Red de interacción proteína-proteína (PPI) de los genes más desregulados en el Grupo 1. La intensidad del color de los nodos refleja el grado de interacción (conectividad), con los nodos más oscuros indicando una mayor conectividad o un estado central dentro de la red. (A) Genes expresados diferencialmente que están relacionados con la señalización proliferativa y el control tumoral. (B) Genes expresados diferencialmente que están relacionados con la Inmunidad Inflamatoria Sistémica. (C) Genes expresados diferencialmente que están relacionados con la Inestabilidad transcriptómica. (D) Genes expresados diferencialmente que están relacionados con Mitocondrial. (E) Genes expresados diferencialmente que están relacionados con la degradación del Proteasoma.
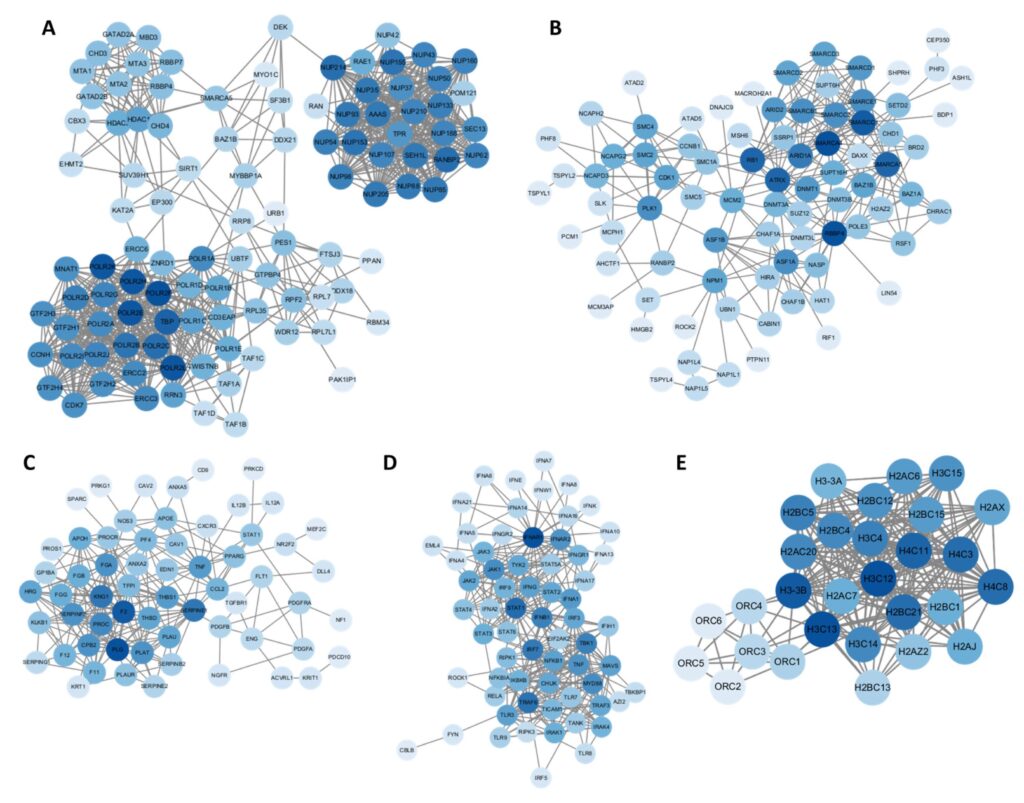
Figura 3. Red de interacción proteína-proteína (PPI) de los genes más desregulados en el Grupo 2. La intensidad del color de los nodos refleja el grado de interacción (conectividad), y los nodos más oscuros indican una mayor conectividad o un estado central dentro de la red. (A) Red de interacción proteína-proteína para los genes sobrerregulados que están relacionados con la inestabilidad transcriptómica, el estrés traslacional. (B) Red de interacción proteína-proteína para los genes regulados al alza relacionados con la inestabilidad genómica y el cambio epigenético. (C) Red de interacción proteína-proteína para los genes corregidos a la baja relacionados con la disfunción endotelial. (D) Red de interacción proteína-proteína para los genes desregulados que están relacionados con la Respuesta Inflamatoria e Inmunitaria Sistémica. (E) Red de interacción proteína-proteína para los genes regulados al alza que están relacionados con la Señalización Proliferativa y el Control Tumoral Suprimido.
4. Discusión […]
5. Conclusiones
Este estudio proporciona pruebas transcriptómicas de alteraciones moleculares en dos poblaciones de pacientes -aquellos con efectos adversos no malignos de nueva aparición y aquellos con cánceres recién diagnosticados- meses a años después de la vacunación con ARNm COVID-19. Mediante análisis de expresión diferencial y GSEA, identificamos firmas distintivas de disfunción mitocondrial, estrés traslacional, desregulación inmunitaria, alteración endotelial y señalización proliferativa en ambas cohortes. En particular, aunque ambos grupos compartían perturbaciones transcripcionales en las vías inmunitaria y traslacional, el grupo de cáncer presentaba firmas adicionales de inestabilidad genómica y remodelación epigenética.
La expresión persistente de la proteína espiga, la actividad sintética prolongada del ARNm y las modificaciones del ARN como la N1-metilpseudouridina parecen contribuir a la actividad ribosómica aberrante sostenida, al estrés de la proteostasis y a la activación inmunitaria. Nuestros hallazgos también ponen de relieve señales transcripcionales indicativas de condiciones promotoras de tumores, incluyendo redes p53 suprimidas, dianas MYC activadas y señalización de interferón alterada, particularmente en el contexto de la desregulación epigenética en la cohorte de cáncer. Estas observaciones sugieren que la reprogramación transcriptómica inducida por la vacuna puede afectar de forma diferencial a los individuos, genética o inmunológicamente, durante un largo periodo de tiempo tras la vacunación.
Referencias
————————-